優(yōu)勢
Arraystar m6A 單堿基分辨率芯片相比于傳統(tǒng)m6A-seq具有優(yōu)勢:
- 利用不依賴m6A抗體富集的方法系統(tǒng)性分析RNA m6A修飾譜
- 單堿基分辨率精確檢測m6ACA修飾位點
- 定量分析m6A修飾的百分比和豐度
- 樣本要求量低,可低至1 ug總RNA
- 多個專業(yè)數(shù)據(jù)庫收集并注釋可量化的單個、多聚和成簇的m6ACA位點
簡介
在單堿基水平上定量m6A修飾仍然具有極大的挑戰(zhàn)性。傳統(tǒng)的m6A/MeRIP-seq這兩種方法使得研究m6A表觀轉(zhuǎn)錄組學(xué)成為可能[1-5],卻不能精確鑒定出MeRIP-seq的peak區(qū)域內(nèi)具體是哪一個腺嘌呤(A)發(fā)生了m6A修飾,也不能定量分析每個位點的修飾百分比[6]。
為了解決這一難題,Arraystar公司研發(fā)了m6A單堿基分辨率芯片,可以將m6A修飾精確定位到具體的腺嘌呤(A)上,并且能定量分析m6A修飾位點的百分比及差異變化。
|
Arraystar m6A 單堿基分辨率芯片 |
MeRIP-Seq |
定量 |
m6A修飾百分比
m6A修飾的RNA豐度
m6A修飾的RNA百分比及豐度的差異變化 |
無m6A修飾百分比
只能提供m6A修飾的RNA豐度變化 |
m6A位點分辨率 |
單堿基水平 |
~ 100 nt |
樣本起始量 |
≥1μg總RNA |
≥120μg總RNA |
Poly(A)富集/rRNA去除 |
不需要 |
需要(甚至需要放大) |
RNA完整性要求 |
要求低 |
要求高 |
不依賴抗體的m6A修飾檢測方法
由于m6A抗體會與其他修飾(比如m6Am)產(chǎn)生交叉反應(yīng)[1, 7-9],所以基于m6A抗體的檢測方法其特異性存在限制。并且,在缺乏正交實驗作為獨立參照的情況下,m6A抗體檢測出的m6A修飾譜無法系統(tǒng)性評估檢測結(jié)果的準確度。與依賴于m6A抗體免疫富集的MeRIP和miCLIP方法不同,Arraystar提出,基于甲基化敏感的RNA內(nèi)切酶MazF的酶切原理設(shè)計出的芯片,可以系統(tǒng)性檢測m6A修飾譜。
單堿基分辨率定位m6A修飾位點
RNA內(nèi)切酶MazF識別RNA單鏈并在非甲基化的ACA位點的5’端進行切割,而不能切割甲基化的m6ACA位點(圖1) [1, 10]。將抽提的總RNA樣本分為兩份,一份不加MazF處理,標記上Cy3熒光,另一份加MazF處理后,標記上Cy5的熒光。標記好后的RNA樣本合并在一起與Arraystar m6A單堿基分辨率芯片雜交,從而定量分析每個位點的m6A修飾百分比和豐度(圖2)。
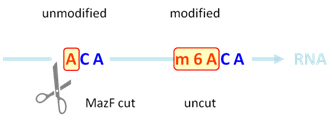
圖 1. MazF可以在非甲基化ACA位點切割,而無法在甲基化m6ACA位點發(fā)揮作用
m6A修飾百分比
m6A修飾百分比(在某個位點處發(fā)生m6A修飾的RNA與未發(fā)生m6A修飾的RNA之間的比值)的檢測,對于闡明m6A修飾的重要功能、回答關(guān)于m6A甲基化的添加/識別/去除、m6A的調(diào)控機制、應(yīng)激過程的m6A修飾動態(tài)變化及m6A修飾位點研究的重要性等問題都是至關(guān)重要的[3, 11, 12]。m6A修飾的百分比可以通過每個位點的雙熒光通道信號值進行定量,解決了長期以來無法檢測m6A修飾百分比動態(tài)變化的困擾。
低樣本量要求
Arraystar m6A單堿基分辨率芯片只需要低至1μg的總RNA。MazF的酶促反應(yīng)即使在ng或pg級別的極低RNA量的情況下,仍然可以保持極高的敏感性和特異性。因此,稀有樣本,尤其是病理切片、活檢樣本、少量分選細胞、小型動物模型,都可以用m6A單堿基分辨率芯片進行m6A修飾的高通量篩選。
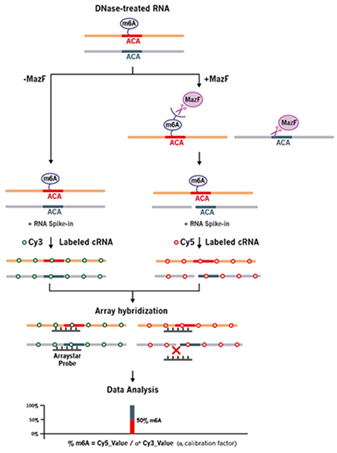
圖 2. Arraystar m6A單堿基分辨率芯片實驗流程
可靠的指標收集和全面的注釋信息
大部分m6A修飾發(fā)生在含有核心ACA序列的保守motif區(qū)域,通常稱為是m6ACA位點。為了收集可靠的m6ACA位點,Arraystar m6A單堿基分辨率芯片設(shè)計了系統(tǒng)性的檢測方法,通過探針對所有的m6ACA位點進行定量分析,最終得到可靠性高的ACA位點。在一個ACA位點的40nt范圍內(nèi)不存在其他ACA位點就可以被一個探針特異性檢測,這個位點被定義為可定量的單一ACA位點。用一個探針檢測到的聚集在20nt范圍內(nèi)的串聯(lián)ACA位點,定義為可定量的多聚ACA位點。另外,當(dāng)多個單一ACA位點或多聚ACA位點分布在500nt范圍內(nèi),并且兩者之間的距離<100nt,他們就會聚集成一個成簇ACA 位點(圖3),這種位點可以通過探針檢測單一ACA位點和多聚ACA位點進行分析。在RNA中,不是所有的ACA序列都有m6A修飾。Arraystar m6A單堿基分辨率芯片中收錄的高可信度與極高可信度的單一、多聚及成簇的可定量的ACA位點均可在miCLIP數(shù)據(jù)集中查找到對應(yīng)m6A修飾位點[13-16],或修飾位點接近于m6A-seq的峰值處[17]。
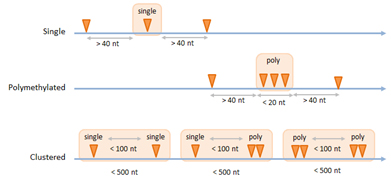
圖 3. 芯片可定量的單一、多聚、成簇m6ACA位點的收集方式。
單一、多聚、成簇m6A修飾位點的重要功能
單一m6A修飾位點通常與mRNA翻譯起始和翻譯延伸的動態(tài)調(diào)控相關(guān),也可以調(diào)控非編碼RNA的降解和活性。
5’UTR區(qū)的單一m6A修飾位點促進HSP70 mRNA非帽子依賴的翻譯起始
Hsp70 mRNA的5’UTR區(qū)只要有一個堿基發(fā)生m6A修飾便足以啟動非經(jīng)典的非帽子依賴的翻譯途徑(圖4) [18]。在壓力條件下,定位在核里的識別蛋白YTHDF2阻止FTO對m6A修飾位點的去甲基化過程。翻譯起始因子eIF3可以直接結(jié)合在m6A位點,促進HSP70 mRNA的翻譯過程。因此,壓力應(yīng)激下,細胞會終止大量的其他mRNA的翻譯過程,但Hsp70 mRNA仍然可以起始非帽子依賴的翻譯途徑。
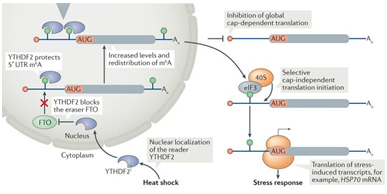
圖 4. 熱休克壓力下,HSP70 mRNA通過單個m6A修飾位點起始非帽子依賴的翻譯途徑的分子機制[18, 19]。
CDS區(qū)的單個m6A修飾位點調(diào)控mRNA翻譯的動態(tài)變化
在mRNA編碼序列區(qū)(CDS)發(fā)生的單個m6A修飾可以調(diào)控翻譯延伸的動態(tài)變化[20]。一個密碼子上發(fā)生m6A修飾將下調(diào)同工tRNA的解碼效率,也可以作為tRNA結(jié)合mRNA與翻譯延伸的阻礙物。m6A阻止翻譯進行,可能會產(chǎn)生截短的蛋白鏈,進而影響正在翻譯的蛋白的折疊、與分子伴侶的互作、定位信號的識別,這些都是導(dǎo)致蛋白命運改變與活性改變的潛在因素[21]。可以想象,如果m6A修飾使得一個密碼子錯譯成其他的氨基酸,即便m6A修飾豐度很低,也將引起蛋白功能缺失的變異,進而引起一系列生物學(xué)效應(yīng)[20](圖. 5)。
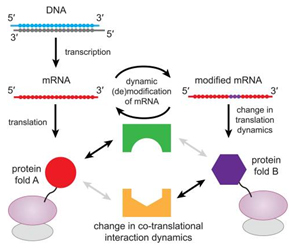
圖. 5. 翻譯延伸的動態(tài)學(xué)調(diào)控可以影響共同翻譯的初生蛋白的折疊,以及與其他伴侶分子的互作[20]。
單一m6A修飾位點調(diào)控lncRNA功能缺失
linc1281的單一m6A修飾位點(A917, A1025和A1056)足以調(diào)控linc1281吸附let-7的能力(圖6) [22]。linc1281的m6A修飾通過阻斷這些干細胞多能性相關(guān)的miRNA的功能,確保小鼠胚胎干細胞的特性。
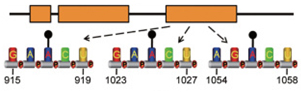
圖6. linc1281最后一個外顯子上的單一m6A修飾位點,調(diào)控linc1281結(jié)合let-7,發(fā)揮ceRNA機制,促進mSEC分化的功能[22]。
多聚與成簇m6A位點參與mRNA相分離
多個m6A位點聚集成簇,對于細胞質(zhì)中mRNA的降解,調(diào)控mRNA半衰期具有重要作用。m6A識別蛋白通過靶向mRNA上的m6A修飾位點進入細胞質(zhì)組分促進細胞相分離調(diào)控mRNA代謝命運。高度m6A修飾的多聚甲基化區(qū)域?qū)ncRNA功能激活也是必不可少的
多聚和成簇(而非單一)的m6A修飾位點,通過調(diào)控相分離的方式,將mRNA劃分至不同的亞細胞組分(圖7) [23]。胞外可自發(fā)進入相分離。多聚甲基化修飾的mRNA為YTHDF的結(jié)合提供多個位點,促使YTHDF將mRNA帶入不同的分離相的細胞組分中,比如P小體、應(yīng)激顆粒、神經(jīng)細胞RNA顆粒,從而介導(dǎo)RNA的不同代謝命運。例如,多聚甲基化的mRNA滯留在應(yīng)激顆粒中可以抑制mRNA的翻譯、YTHDF2識別多聚甲基化mRNA進入P小體可以促進mRNA的降解[24]。然而,單一的m6A甲基化位點在此過程中不足以提供YTHDF結(jié)合的親和力[23]。
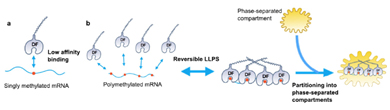
圖7. 多聚m6A甲基化位點結(jié)合胞質(zhì)中的YTHDF1/2/3識別蛋白時具有多重的親和力,并被分離至亞細胞組分調(diào)控RNA代謝命運。
m6A 表觀轉(zhuǎn)錄組學(xué)研究路線圖
Arraystar m6A 單堿基分辨率芯片詳情
| |
Human |
Mouse> |
Rat |
| 芯片規(guī)格 |
8 x 15K |
8 x 15K |
8 x 15K |
| 探針總數(shù) |
14,321 |
14,319 |
14,581 |
| 探針長度 |
60nt
|
| m6ACA位點來源 |
miCLIP dataset[13-16]; m6A-seq peak summits[17] |
| 單一m6ACA 位點 |
11,237 |
11,120 |
11,499 |
| 多聚m6ACA 位點 |
3,084 |
3,199 |
3,082 |
| 成簇m6ACA 位點 |
693 |
279 |
2,614 |
References
[1] Garcia-Campos M A, Edelheit S, Toth U, et al. Deciphering the "m(6)A Code" via Antibody-Independent Quantitative Profiling[J]. Cell, 2019,178(3):731-747.
[2] Knuckles P, Buhler M. Adenosine methylation as a molecular imprint defining the fate of RNA[J]. FEBS Lett, 2018,592(17):2845-2859.
[3] Schwartz S. Cracking the epitranscriptome[J]. RNA, 2016,22(2):169-174.
[4] Meyer K D, Jaffrey S R. Rethinking m(6)A Readers, Writers, and Erasers[J]. Annu Rev Cell Dev Biol, 2017,33:319-342.
[5] Yue Y, Liu J, He C. RNA N6-methyladenosine methylation in post-transcriptional gene expression regulation[J]. Genes Dev, 2015,29(13):1343-1355.
[6] Liu N, Parisien M, Dai Q, et al. Probing N6-methyladenosine RNA modification status at single nucleotide resolution in mRNA and long noncoding RNA[J]. RNA, 2013,19(12):1848-1856.
[7] Dominissini D, Moshitch-Moshkovitz S, Schwartz S, et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq[J]. Nature, 2012,485(7397):201-206.
[8] Schwartz S, Bernstein D A, Mumbach M R, et al. Transcriptome-wide mapping reveals widespread dynamic-regulated pseudouridylation of ncRNA and mRNA[J]. Cell, 2014,159(1):148-162.
[9] Linder B, Grozhik A V, Olarerin-George A O, et al. Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome[J]. Nat Methods, 2015,12(8):767-772.
[10] Imanishi M, Tsuji S, Suda A, et al. Detection of N(6)-methyladenosine based on the methyl-sensitivity of MazF RNA endonuclease[J]. Chem Commun (Camb), 2017,53(96):12930-12933.
[11] Grozhik A V, Jaffrey S R. Distinguishing RNA modifications from noise in epitranscriptome maps[J]. Nat Chem Biol, 2018,14(3):215-225.
[12] Meyer K D, Jaffrey S R. The dynamic epitranscriptome: N6-methyladenosine and gene expression control[J]. Nat Rev Mol Cell Biol, 2014,15(5):313-326.
[13] Linder B. et al. (2015) Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome. [PMID: 26121403]
[14] Ke S. et al. (2015) A majority of m6A residues are in the last exons, allowing the potential for 3' UTR regulation. [26404942]
[15] Chen K. et al. (2015) High-Resolution N6-Methyladenosine (m6A) Map Using Photo-Crosslinking-Assisted m6A Sequencing. [25491922]
[16] Kai X. et al. (2017) Mettl3-mediated m6A regulates spermatogonial differentiation and meiosis initiation. [28809392]
[17] Schraga Schwartz, et al. (2013) High-Resolution Mapping Reveals a Conserved, Widespread, Dynamic mRNA Methylation Program in Yeast Meiosis. [24269006]
[18] Zhou, J., et al. (2015) "Dynamic m(6)A mRNA methylation directs translational control of heat shock response" Nature 526(7574):591-4 [PMID: 26458103]
[19] Kathrin Leppek, et al. (2018) Functional 5′ UTR mRNA structures in eukaryotic translation regulation and how to find them. [PMID: 29165424]
[20] Choi, J., et al. (2016) "N(6)-methyladenosine in mRNA disrupts tRNA selection and translation-elongation dynamics" Nat Struct Mol Biol 23(2):110-5 [PMID: 26751643]
[21] Kim, S. J., et al. (2015) "Protein folding. Translational tuning optimizes nascent protein folding in cells" Science 348(6233):444-8 [PMID: 25908822]
[22] Yang, D., et al. (2018) "N6-Methyladenosine modification of lincRNA 1281 is critically required for mESC differentiation potential" Nucleic Acids Res 46(8):3906-3920 [PMID: 29529255]
[23] Ries, R. J., et al. (2019) "m(6)A enhances the phase separation potential of mRNA" Nature 571(7765):424-428 [PMID: 31292544]
[24] Wang, X., et al. (2014) "N6-methyladenosine-dependent regulation of messenger RNA stability" Nature 505(7481):117-20 [PMID: 24284625]
康成生物丨數(shù)譜生物獨家提供技術(shù)服務(wù)
康成生物丨數(shù)譜生物國內(nèi)獨家提供Arraystar m6A單堿基分辨率芯片全程優(yōu)質(zhì)技術(shù)服務(wù),歡迎咨詢。
|
|
|
