聚焦品牌優(yōu)勢
 |
基于circRNA研究領(lǐng)域頂尖期刊和權(quán)威公共數(shù)據(jù)庫,運用強大的信息篩選系統(tǒng),挑選出可靠的circRNA。 |
 |
剪接位點特異性探針與Rnase R預(yù)處理雙重保障,即便在相應(yīng)線性RNA存在的情況下也能夠特異性檢測circRNA。 |
 |
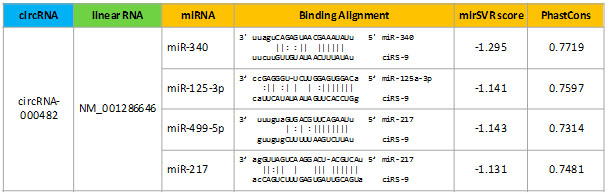
數(shù)據(jù)報告提供circRNA可能結(jié)合的miRNA以及對應(yīng)的host gene和線性RNA,便于從不同角度研究circRNA的作用機制。 |
 |
circRNA的特征決定了通過高通量測序的方法檢測結(jié)果不準(zhǔn)確,Arraystar circRNA芯片可以恰到好處地破解這個難題。 |
circRNA介紹
circRNA是一類由特殊剪接機制形成的、具有閉合環(huán)狀結(jié)構(gòu)的非編碼RNA,也是目前生命科學(xué)和醫(yī)學(xué)領(lǐng)域的研究熱點分子。circRNA分子的組織特異性、疾病特異性、時序特異性及高穩(wěn)定性等特征,使得circRNA作為臨床疾病的biomarker具有明顯的優(yōu)勢。circRNA作為相應(yīng)線性RNA的補充,表達量水平高,可以在細胞核中穩(wěn)定存在并在細胞質(zhì)中富集;circRNA具有大量miRNA結(jié)合位點,由于不完全配對,故不會被miRNA靶定降解;由于這些特征,circRNA被公認為高效的天然miRNA海綿,在基因調(diào)節(jié)中發(fā)揮競爭性內(nèi)源RNA(ceRNA)作用。此外,一些內(nèi)含子類型的circRNA會促進宿主基因轉(zhuǎn)錄。生物學(xué)功能強大的circRNA分子猶如一座亟待開發(fā)的“金礦”。 為了能夠幫助科研工作者對circRNA進行更好地研究,美國Arraystar公司在其首款circRNA芯片基礎(chǔ)上迅即升級版本為V2.0,以便于研究者對不同生理和病理狀態(tài)下對 circRNA的表達變化進行系統(tǒng)的研究。
circRNA競爭性吸附miRNA
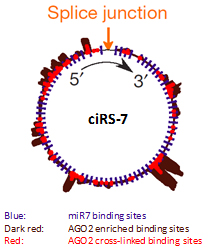
circRNA序列上含有大量的miRNA結(jié)合位點,當(dāng)circRNA與miRNA同處于細胞漿時會引起circRNA對miRNA的吸附(miRNA sponge)。例如,通過PAR-CLIP實驗證實了ciRS-7與miR-7以及RNA誘導(dǎo)沉默復(fù)合體(RISC)重要組成成分AGO2蛋白結(jié)合;對生物素偶聯(lián)的miRNA進行pull down實驗,進一步驗證了ciRS-7吸附miR-7功能的存在。(圖1)
|
圖1. ciRS-7序列上含有七十多個miR-7結(jié)合位點,它可與miR-7以及AGO2蛋白結(jié)合。 |
circRNA調(diào)節(jié)RNA結(jié)合蛋白(RBP)
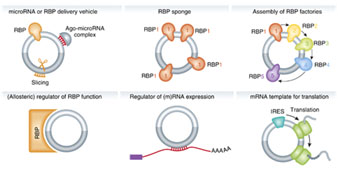
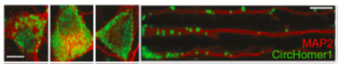
circRNA可以與多種RNA結(jié)合蛋白(RBP)作用,進而發(fā)揮相應(yīng)的分子生物學(xué)功能(圖2);例如circRNA可以充當(dāng)RBP的亞細胞轉(zhuǎn)移或定位的載體(圖3)、作為RBP的分子海綿,組裝RBP復(fù)合物以及作為酶活性調(diào)節(jié)的輔因子等。Antisense類型的circRNA還可以通過堿基互補配對的方式與相應(yīng)的mRNA結(jié)合,進行調(diào)控mRNA的穩(wěn)定性。
|
圖2. circRNA與RNA結(jié)合蛋白(RBP)之間的相互作用 |
|
|
|
圖3. circRNA在神經(jīng)元胞體及樹突動態(tài)分布示意圖 |
circRNA的生物學(xué)功能
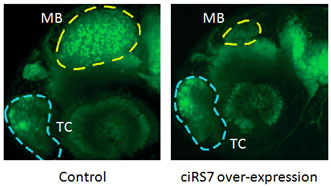
miRNA分子調(diào)控生物體內(nèi)1/3左右的基因,circRNA對miRNA的吸附會影響基因的表達,進而引起生物學(xué)表型或狀態(tài)的改變(圖4)。相當(dāng)大一部分環(huán)狀RNA的靶標(biāo)基因是介導(dǎo)信號傳導(dǎo)的蛋白激酶。研究還表明,circRNA的異常表達往往和臨床疾病相關(guān),比如腫瘤、阿爾茨海默癥、動脈粥樣硬化等。
|
圖4. 斑馬魚胚胎過表達ciRS-7(右圖)會導(dǎo)致中腦(MB)縮小,而端腦(TC)大小不受影響。 |
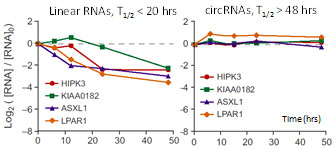
circRNA的高穩(wěn)定性
circRNA分子獨特的閉合環(huán)狀結(jié)構(gòu),不受核酸外切酶影響,使circRNA的穩(wěn)定性要遠遠高于同一個基因轉(zhuǎn)錄的相應(yīng)線性RNA分子(圖5)。circRNA的高穩(wěn)定性預(yù)示其非常適用于臨床biomarker研究。
|
圖5. circRNA在細胞內(nèi)穩(wěn)定性遠高于同一個基因轉(zhuǎn)錄的相應(yīng)線性RNA,前者半衰期>48h(右圖),而后者半衰期<20h(左圖)。 |
circRNA高通量篩選的利器:RNA-seq or microarray?
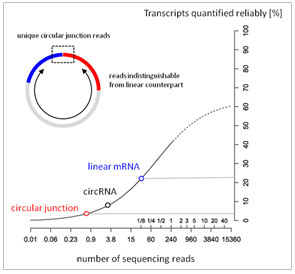
circRNA在細胞內(nèi)含量極低,約占線性RNA的5-10%,通過測序獲得的circular junction reads 數(shù)目更低。RNA測序僅僅能有效檢測低于5% 的circRNAs(圖6),即便將測序量提高到300M reads,對于檢測準(zhǔn)確度的影響也是微乎其微。
測序不是circRNA高通量篩選的優(yōu)質(zhì)檢測手段,circRNA-seq需要雙端模式和極高的測序量。此外,后續(xù)的數(shù)據(jù)分析(如mapping所需數(shù)據(jù)庫、circRNA轉(zhuǎn)錄本組裝、特殊生物信息學(xué)算法、計算機篩選程序等)也存在一系列難題。由于上述因素所限,通過circRNA-seq發(fā)現(xiàn)新的circRNA的可能性幾乎為零。
circRNA芯片采用特異性剪接位點探針與外切酶預(yù)處理雙重保障,能夠準(zhǔn)確檢測樣本中circRNA的表達。芯片雜交不受RNA豐度的影響,即便在一個細胞中只含一個circRNA拷貝也能夠做到特異性檢測。此外,芯片避免了circRNA-seq復(fù)雜的建庫過程,其高效成熟的技術(shù)是circRNA高通量篩選的優(yōu)質(zhì)平臺。
|
圖6. 定量可靠的轉(zhuǎn)錄本比例與測序深度關(guān)系圖。RNA測序一般產(chǎn)生低于30M mRNA reads(藍圈),低于0.5M circular junction reads(紅圈),低于5%的circular junction真正得到有效檢測。(Adopted from Labaj et al, (2011) Bioinformatics [PMID 21685096].) |
國際尖端circRNA研究利器
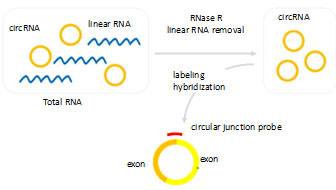
Arraystar circRNA芯片是準(zhǔn)確檢測樣本中circRNA含量理想的工具。剪接位點特異性探針與Rnase R預(yù)處理雙重保障,即便在相應(yīng)線性RNA存在的情況下也能夠做到特異性檢測circRNA。數(shù)據(jù)報告中詳盡的circRNA注釋信息十分便于客戶進行circRNA機制研究。
|
圖7. circRNA芯片實驗大體流程(上圖)及詳細的circRNA注釋(下圖): circRNA對應(yīng)的線性RNA及miRNA的結(jié)合位點。 |
研究技術(shù)路線
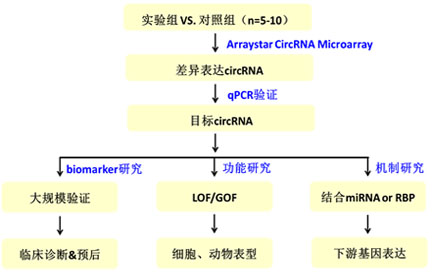
通過芯片篩選獲得組間差異表達的circRNA,接下來通過低通量circRNA-PCR的驗證(圖8)鎖定目標(biāo)circRNA分子。目標(biāo)circRNA分子可以展開不同方向的研究:臨床biomarker或功能機制(圖9)。
|
|
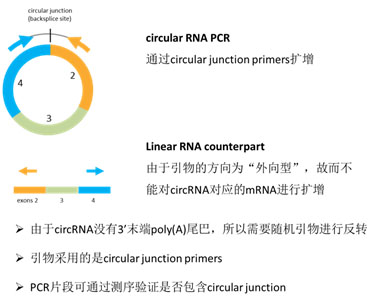
圖8. circRNA PCR驗證引物設(shè)計示意圖 |
圖9. circRNA基本研究思路示意圖 |
環(huán)狀RNA芯片參數(shù)
|
Human |
Mouse |
Rat |
| Total Number of unique circRNAs |
13,617 |
14,236 |
14,145 |
| Probe Length |
60nt |
60nt |
60nt |
| Probe Region |
Circular junctions of circRNA |
| Probe Specificity |
Transcript-specific |
| circRNA Enrichment |
RNase R treatment |
| Labeling Method |
Labeling by random primers |
| Circular RNA Sources |
| Salzman’s circRNAs (2013) |
8,529 |
|
|
| Memczak’s circRNAs (2013) |
1,601 |
1,750 |
|
| Zhang’s circRNAs (2013) |
93 |
|
|
| Zhang’s circRNAs (2014) |
4,980 |
|
|
| Jeck’s circRNAs (2014) |
3,769 |
|
|
| Guo’s circRNAs (2014) |
5,536 |
570 |
|
| You's circRNAs (2015) |
|
13,300 |
12,298 |
| Mouse circRNA orthologs |
|
|
1,668 |
| Human circRNA orthologs |
|
|
179 |
| Array Format |
8 * 15K |
8 * 15K |
8 * 15K |
參考文獻
[1] Guo, J. U., V. Agarwal, et al. (2014). "Expanded identification and characterization of mammalian circular RNAs." Genome Biol 15(7): 409.
[2] Jeck, W. R., J. A. Sorrentino, et al. (2013). "Circular RNAs are abundant, conserved, and associated with ALU repeats." RNA 19(2): 141-157.
[3] Memczak, S., M. Jens, et al. (2013). "Circular RNAs are a large class of animal RNAs with regulatory potency." Nature 495(7441): 333-338.
[4] Salzman, J., R. E. Chen, et al. (2013). "Cell-type specific features of circular RNA expression." PLoS Genet 9(9): e1003777.
[5] Zhang, X. O., H. B. Wang, et al. (2014). "Complementary sequence-mediated exon circularization." Cell 159(1): 134-147.
[6] Zhang, Y., X. O. Zhang, et al. (2013). "Circular intronic long noncoding RNAs." Mol Cell 51(6): 792-806.
[7] You X. et. al. (2015) “Neural circular RNAs are derived from synaptic genes and regulated by development and plasticity.” Nat Neurosci.
2015 Apr;18(4):603-10
[8] Hentze and Preiss (2013) “Circular RNAs: splicing's enigma variations.” EMBO J 3;32(7):923-5.
康成生物是Arraystar公司在中國地區(qū)的唯一代理商,提供從樣品到數(shù)據(jù)分析的全程技術(shù)服務(wù)。
|